Einführung
Einführung
Dieser Tage hatte ich das Vergnügen am Biozentrum der Universität Köln quasi ein "Praktikum" zur Gen-Sequenzierung der Ciliaten (Wimpertiere) absolvieren zu können. Mit Kollegen aus der Biologie haben wir ca. 20 Stämme meiner gesammelten Spezies und weiterer Samples für eine Gen-Sequenzierung vorbereitet, deren Proben ich sammelte und gemeinsam mit einigen befreundeten Mikroskopikern um weitere Spezies vervollständigte. Um es vorweg zu nehmen, ich habe die "Prüfung" bestanden und die Kollegen haben sehr wertvolle Beiträge geliefert. Alles in allem bis heute ein sehr erfolgreiches Projekt mit ausgebildeten Biologen und Citizen Science! Wir haben eine Reihe von sehr guten Gen-Sequenzen erhalten für die weitere Auswertung einer bestimmten Gattung von Wimpertieren. Zu gegebener Zeit werde ich hierüber mehr berichten.
Die Gen-Sequenzierung der DNA von Ciliaten ergänzt neben der traditionellen Versilberung heute die Determinierung von Arten. Dabei ist es durchaus nicht so, dass man die Arten nur mit Hilfe der DNA Analyse bestimmen könnte. Vielmehr unterstützt die DNA Analyse die taxonomische Einordnung der geschätzt wenigstens 8.000 Arten dieser einzelligen Lebewesen. Hat man die Arten einmal taxonomisch eingeordnet, so helfen in vielen Fällen auch lichtmikroskopische Befunde die Art zu bestimmen.
DNA Vervielfältigung
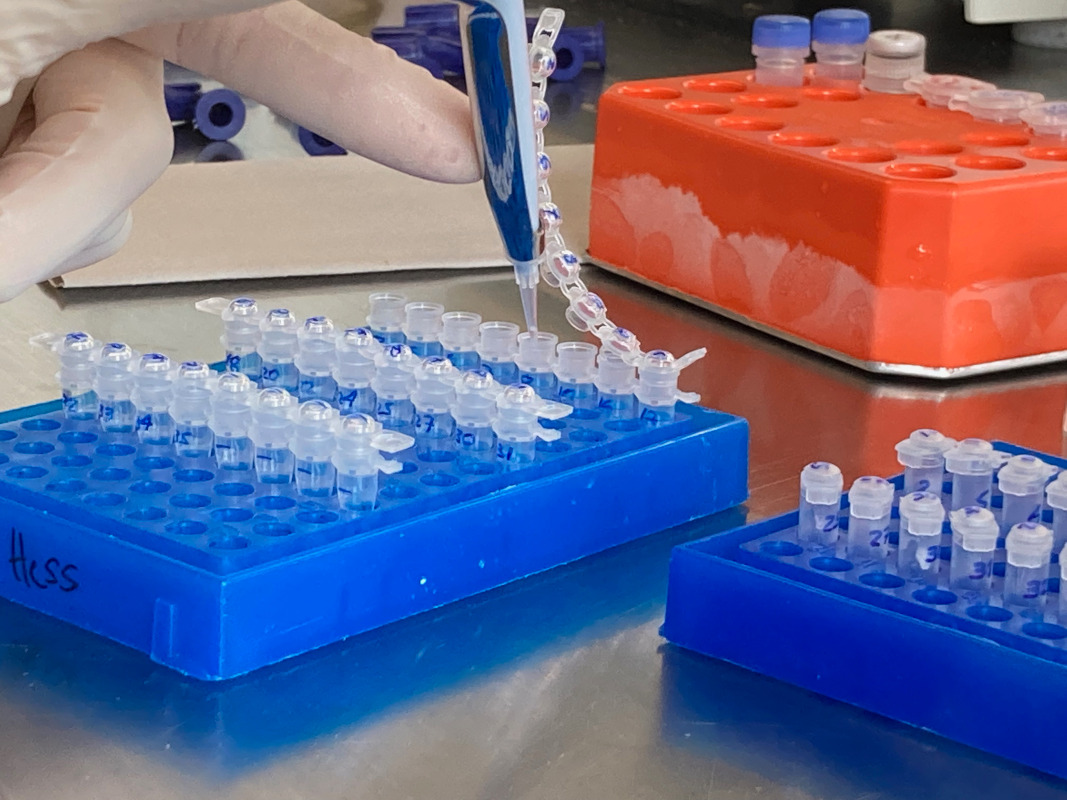
Die ersten Schritte der Gen-Sequenzierung betreffen die Probenvorbereitung und die Vervielfältigung der DNA. Wir haben Single-Cell DNA Analysen durchgeführt. Hierzu pickt man ein einzelnes Wimpertier unter dem Mikroskop heraus und reinigt es anschließend in DNA-freiem, ultrareinem Wasser ein paar Mal. Anschließend wird die DNA extrahiert und in einem weiteren Schitt vervielfältigt. Hierfür wendet man ein spezielles Protokoll an, um die Polymerase-Kettenreaktion, kurz PCR, in einem Thermocycler durchzuführen.
Die PCR vervielfältigt die DNA soweit, dass man diese mit statistischen Verfahren sequenzieren kann. Dies geschieht mit einer modernen Variante der Sanger-Sequenzierung. Bevor die vervielfältigte DNA zur Sequenzierung gegeben wird, erfolgt eine Endkontrolle der vervielfältigten DNA. Man erstellt einen sogenannten Gel-Blot. In einer Elektrophoresekammer wird ein Gel aus Agarose ausgegossen, das mit einem Fluorochrom eingefärbt ist. Kleine Kämmchen sind hier eingehängt, die nach dem Aushärten des Agarose-Gels winzige Fächer im Gel ergeben. Von der vervielfältigten DNA entnimmt man eine kleine Menge und färbt sie mit einer Farbstoff-Pufferlösung an, damit man besser sieht, wohin man sie pipettiert und wie weit die Elektrophorese fortgeschritten ist. Diese gefärbte DNA wird anschließend in die kleinen Fächer des Gels pipettiert.
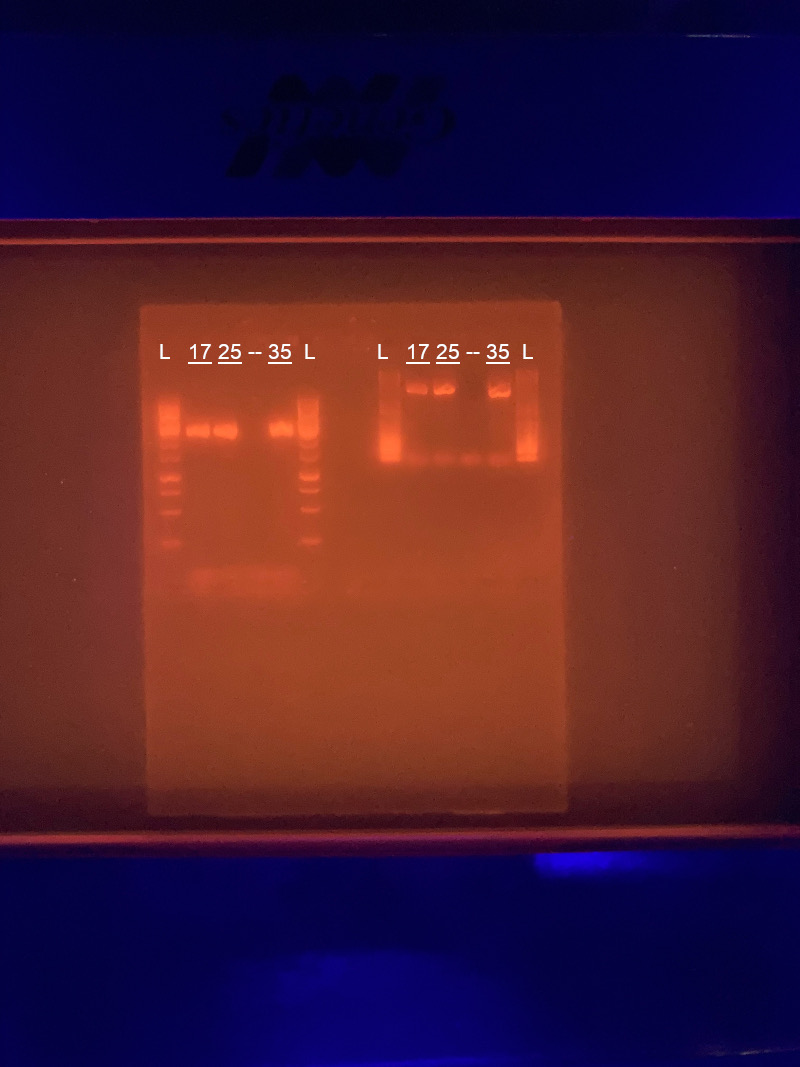
Mit Hochspannung (ca. 100 V) wird die DNA im elektrischen Feld nun durch das Gel gezogen. Dabei wandern die verschieden langen DNA-Fragmente unterschiedlich weit. Die kürzeren an vorderster Front, die längeren werden im Gel stärker ausgebremst. So ergeben unterschiedlich lange DNA Stänge ein Linienmuster mit unterschiedlicher Lage der Stränge. Zum Vergleich benutzt man eine DNA-Leiter. Dies ist eine Mischung aus synthetischer DNA mit genau definierter Länge. So kann man die Länge der erhaltenen DNA-Fragmente einschätzen. Nach einer Weile wird die Elektrophorese gestoppt und man betrachtet das Gel anschließend in einem Illuminator. Hier kommt nun das Fluorochrom im Gel zum Einsatz. Dieser Farbstoff bindet spezifisch an die DNA und ergibt dort, wo sich nun die DNA-Stränge befinden markante Linien im Gel. Mit kurzwelligem Licht angeregt, wird die DNA aufleuchten.
Endkontrolle passiert, die PCR ist hier geglückt. Die Beschriftung in meiner Abbildung bezeichnet verschiedene DNA Proben, wobei die Zahlen verschiedene Stämme von Ciliaten aus meinen Kulturen bezeichnen und "L" die DNA-Leiter. Den selbst angefertigten Gelblock habe ich in einer Kunststofffolie gesichert und mit nach Hause genommen. Frei nach dem Motto, kein Gel-Blot ist unnütz, man kann ihn immer noch als schlechtes Beispiel verwenden.
Do-It-Yourself Illuminator
Ich hatte mir vor einer Weile einen eigenen LED-Beleuchtungskasten für die Illuminierung von Gel-Blots gebaut, um später DNA Proben im eigenen Labor vorzubereiten. DIY-Bio ist das Motto von heute und Molekularbiologie macht heute schließlich jeder in seiner eigenen Küche. Also kann das ein gestandener Physiker erst recht. Heute wird die Box erstmals getestet.
Mein eigener Illuminator wurde mit Komponenten aus dem Baumarkt und vom lokalen Elektronikhändler gebaut. Eine Hand voll leuchtkräftiger, blauer Nichia LEDs wurden auf einer Platine verlötet und in passende Bohrungen eines U-Profils aus Aluminium gesetzt. Ein regelbares Netzteil dient zur Stromversorgung dieser Beleuchtung mittels Konstantstrom. Die umgebende Holzbox wurde aus MDF-Platten gefertigt. Sie ist von der Dimension her genau auf meine neu erworbene Elektrophoresekammer zugeschnitten. Die LED Schienen sind seitlich in die Innenwand der Box geschraubt, so dass die LEDs genau auf der Höhe des Gels in der Elektrophoresekammer liegen, um diese seitlich zu beleuchten. Schwarze Klebefolie und zwei Tragegriffe geben der innen und außen schwarz lackierten Box ein gefälligeres Aussehen. Das Loch oben dient dazu mit der Kamera Photos von den Gel-Blots zu machen.
Ein Orangefilter dient zum Nachweis der Fluoreszenz der mit einem speziellen DNA-Fluorochrom angefärbten DNA. Dieser Filter für die S/W Fotografie lässt nur Licht ab einer bestimmten Wellenlänge passieren, hier grüne bis rote Farben. Der Filter wird auf das Objektiv der Kamera geschraubt. Dann setzt man die Kamera oben auf das Loch. Die Höhe der Box ist genau so dimensioniert, dass ich mit der Canon Kamera und einem Zoom-Objektiv die Gele dokumentieren kann.
"First-Light" nennt es der Astronom, wenn ein neues Instrument zum ersten Mal das Licht sieht. Für meinen ersten Test will ich die Fotos, wie gestern auch, mit dem iPhone fotografieren. So stelle ich vergleichbare Bedingungen der Beleuchtung her im Vergleich mit dem professionellen Illuminator aus dem Labor der Uni, mit dem ich gestern die Abbildung des Gels aufnahm. Da die Kamera des iPhone kleiner ist, kommt noch ein kleines Stück schwarzen Kartons auf den Filter, um Streulicht zu vermeiden.
Fluoreszenz geht nicht nur mit dem Mikroskop, sondern ist auch sonst ein sehr beliebtes Mittel, bereits geringste Spuren gefärbter DNA nachzuweisen. Meine DNA Illuminierungs-Box ist sicherlich preiswerter, als die professionellen Geräte. Doch heute muss sie endlich zeigen, was sie auf, pardon, in dem Kasten hat.
Das Prinzip ist einfach: Die LED-Schienen beleuchten das Gel mit blauem Licht von der Seite her, die gefärbte DNA fluoresziert grün oder rötlich, je nach verwendetem Farbstoff. Daher kann man Anregungsfarbe und emittiertes Licht gut mit einem Farbfilter voneinander trennen. Der fertige Gel-Block von gestern dient hier als Ersatz für das Gel, das sich später in der beleuchteten Elektrophorese-Kammer befinden wird. Diese Abbildung zeigt die ungefilterte, blaue Beleuchtung des Gels, welches hier nun in meiner eigenen Elektrophoresekammer liegt.
Mit dem Farbfilter auf der Box kann nun die fluoreszierende DNA mit dem Mobiltelefon aufgenommen werden. Der Filter unterdrückt das blaue Anregungslicht. Langwelliges Licht von grün bis rot bleibt sichtbar, das blaue Licht ist unterdrückt. Mein Ergebnis kann sich durchaus sehen lassen. Mit dem aufgelegten Orangefilter sind sowohl die rot gefärbte DNA-Leiter, als auch die Proben selbst hervorragend sichtbar und können sogar mit einem Mobiltelefon gut fotografiert werden. Für die Begutachtung und Dokumentation des Erfolgs oder Misserfolgs einer PCR genügt das allemal. Wir machen hier ja keine High-End Fluoreszenzaufnahmen mit dem Mikroskop, sondern wollen nur einen Gel-Blot begutachten.
Auch hier gilt: Optimieren geht immer. Ein Orangefilter, der später im roten Spektrum einsetzt wird den Rest des grünen Lichts der LEDs besser unterdrücken, um einen besseren Kontrast zu erhalten. Hier sollte man beim roten Fluorochrom zur Markierung der DNA bleiben. Der verwendete Orangefilter lässt grünes bis rotes Licht passieren, so dass auch grüne Fluorochrome verwendet werden können. Leider sind blaue LEDs nicht monochrom, sondern sie emittieren neben der Hauptfarbe anteilig einen doch erheblichen Teil langwelligen, grünen bis gelben Lichts. Dieses Licht dürfte eine schwächere grüne Fluoreszenz überlagern und einen geringeren Kontrast ergeben. Ein zusätzlicher Blaufilter aus einer Filterfolie könnte helfen diese langwelligen Anteile der LEDs abzuschwächen, um die eigentlich grün emittierenden Fluorochroms besser zu kontrastieren. Dies werde ich zu einem späteren Zeitpunkt experimentell überprüfen.
Jedoch: Das Prinzip ist gezeigt, mein selbstgebauter Illuminator funktioniert schon recht ordentlich im Vergleich mit dem Profigerät der Mikrobiologen. Operation geglückt! Der Illumination späterer DNA Präparate mit der eigenen Elektrophoresekammer steht nun nichts mehr im Wege.